Entwicklung und Etablierung von Diagnosemethoden für den zukunftsorientierten Nachweis von Pflanzenviren/Viroide (VirusDetect)
Ziel des Forschungsprojektes ist es, Diagnoseprotokolle für eine vereinfachte, schnellere und auch kostengünstigere Diagnose von bereits bekannten, aber vor allem von neu auftretenden Schaderregern an Kulturpflanzen zu ermöglichen. Dabei sollen zwei PCR-basierte Diagnosemethoden weiterentwickelt und für die Routinetestung etabliert werden.
Einleitung
In der Landwirtschaft können Virusinfektionen bei Pflanzen zu massiven Ertragseinbußen und damit zu gravierenden finanziellen Verlusten führen. Der labordiagnostische Nachweis von Pflanzenkrankheiten ist für eine gezielte Bekämpfungsstrategie unerlässlich. Dies ist insbesondere im Bereich der phytopathogenen Virologie wichtig, da eine rein visuelle Diagnose oft nicht ausreicht. Zudem steht die Landwirtschaft in Zeiten veränderter klimatischer Bedingungen und zunehmender Globalisierung vor immer neuen Herausforderungen in Bezug auf phytopathogene Erreger. Dies bedeutet auch, dass Diagnoselaboratorien neue und moderne Methoden für einen zuverlässigen Nachweis von Krankheitserregern entwickeln müssen, um den gesetzlichen Anforderungen und den Bedürfnissen der Landwirtschaft gerecht zu werden.
Derzeit werden an der LfL Pflanzenviren einzeln getestet, indem für jede Probe ein Test auf ein bestimmtes Virus durchgeführt wird, was viele Ressourcen in Anspruch nimmt und zu langen Wartezeiten führen kann. Zudem erfordert es immer wieder neue Nachweisverfahren, um neue oder unbekannte Viren sicher nachzuweisen. Um diese Probleme zu entschärfen, soll in diesem Projekt eine molekularbiologische Methode entwickelt und so erweitert werden, dass sie für die Routinediagnostik eingesetzt werden kann (generischer Nachweis). Darüber hinaus soll eine bestehende Methode weiterentwickelt werden, die es ermöglicht, mehrere Erreger in einem Analyseansatz zu detektieren (Multiplexing).
Ziele
Hauptziel des Projekts ist es, die Diagnostik im Bereich Pflanzenvirologie effizienter zu gestalten und die Möglichkeit zu schaffen, neue, bisher unbekannte oder neu auftretende Pflanzenviren zu testen.
Generischer Nachweis
Bei der generischen Nachweismethode werden Viren auf Gruppenebene nachgewiesen. Als Grundlage für den molekularbiologischen Nachweis werden hochkonservierte Bereiche im Genom verwendet, die bei möglichst vielen Vertretern der eng miteinander verwandten Viren einer Gruppe auftreten. Anhand der Informationen über die hochkonservierten Genombereiche der Viren in der Virusgruppe werden Primer abgeleitet und entwickelt, die für eine NachweisreaktionPolymerase-Chain-Reaction, PCR eingesetzt werden.
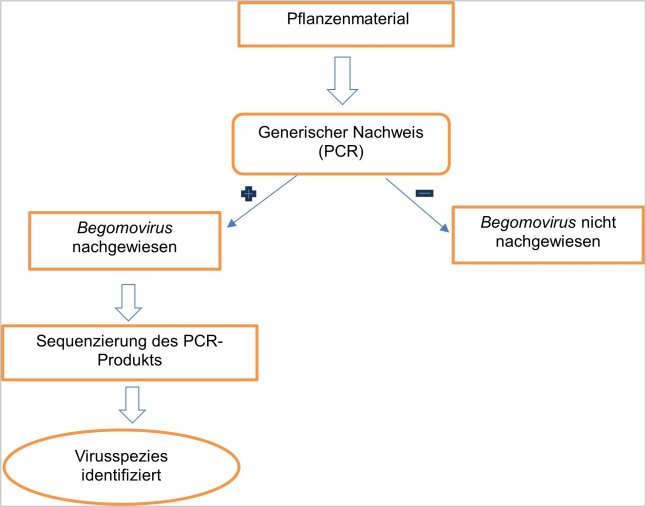
Abbildung 2: Schematische Darstellung einer generischen Virus-Detektion
Nachfolgend an die PCR-Reaktion kann bei einem positiven Ergebnis eine Sequenzierung durchgeführt werden, um die genaue Virusspezies zu identifizieren. Der Fokus der zu entwickelnden Diagnoseverfahren soll dabei auf Virusgruppen liegen, die einen hohen wirtschaftlichen Schaden anrichten können und als Quarantäneschädlinge geführt werden. Zudem soll der generische Nachweis in Zukunft auch im Falle von unbekannten und neu auftretenden Viren für eine gesicherte Nachweisreaktion herangezogen werden.
Unterziel
Multiplex-PCR
Eine weitere Methode, die in diesem Projekt entwickelt werden soll, ist die Multiplex-PCR. Dabei werden mehrere Einzelanalysen in einem einzigen Reaktionsansatz gebündelt, um den Durchsatz der Nachweise zu erhöhen. Ziel ist es, die einzelnen Erregernachweise so zu kombinieren, dass mehrere für diese Wirtspflanze relevante virale Erreger in einem Reaktionsansatz nachgewiesen werden können.
Zum Beispiel: Tomato brown rugose fruit virus (ToBRFV), Pepino mosaic virus (PepMV), Tomato mosaic virus (ToMV), oder Tomato leaf curl New Dehli Virus (ToLCNDV) sind relevante Viren, die Tomaten befallen können und einen hohen wirtschaftlichen Schaden anrichten können. Ziel ist es, diese Viren möglichst in einem Reaktionsansatz mit einer molekularbiologischen Methode wie der konventionellen PCR und qRT-PCR nachzuweisen.

Abbildung 3: Symptombilder einer mit Tomato brown rugose fruit virus (ToBRFV) infizierten Tomatenprobe (Frucht) und Paprikaprobe (Blatt)
Dafür müssen passende Einsatzfelder und mögliche Virus-Kombinationen, die in der Routinediagnostik oder im Rahmen staatlicher Aufgaben sinnvoll kombiniert werden können, identifiziert werden. Insbesondere im Bereich der staatlichen Aufgaben werden Wirtspflanzen auf das Vorhandensein verschiedener viraler Schaderreger untersucht. Hier wäre das Ziel, eine Anwendung zu entwickeln, die mit wenigen Analysen die meisten oder alle in Frage kommenden Viren abdeckt, um Zeit und Kosten zu sparen.
Bei der klassischen PCR werden die Produkte über eine elektrophoretische Auftrennung spezifisch nach der Produktlänge aufgetrennt und geben dadurch Aufschluss über die zu ermittelnde Virusspezies. Bei einer RT-qPCR werden für die Auswertung optische Signale, die bei der Vervielfältigung der entsprechenden Zielsequenzen produziert werden, herangezogen.
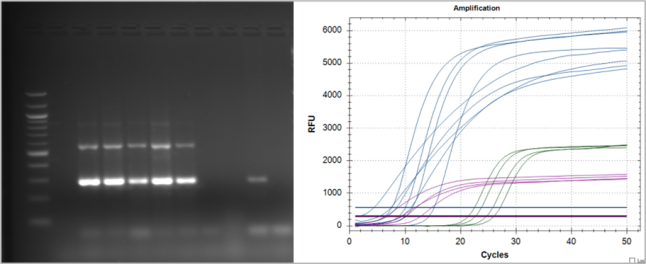
Abbildung 4: Exemplarische Ergebnisse einer klassischen PCR (links) und einer RT-qPCR (rechts)
Methoden
Für beide Testansätze ist vor der Durchführung der Laboruntersuchungen eine ausführliche Literaturrecherche und -analyse erforderlich. Im Falle der generischen Detektion müssen die Virusgruppen identifiziert werden, für die in Datenbanken ausreichende Sequenzinformationen vorliegen, um konservierte Regionen abzuleiten. Auf der Grundlage dieser Informationen können Primer und Protokolle für die durchzuführenden Labortests entwickelt werden. Für das Multiplexverfahren muss im Vorfeld der Laboruntersuchungen analysiert werden, welche Erreger in einem Ansatz diagnostiziert werden sollen, ob entsprechende Protokolle für den Einzelnachweis vorhanden sind und wie diese in einem Ansatz kombiniert werden können. Diese müssen dann in der Praxis auf deren Tauglichkeit getestet und eventuell angepasst werden.
Bei beiden Methoden werden zusätzlich zur Anwendbarkeit und Funktionalität der Methode verschiedene Qualitätssicherungsparameter wie analytische Empfindlichkeit, Selektivität, Reproduzierbarkeit, Robustheit und Wiederholbarkeit umfassend geprüft.
Ergebnisse
Die Auswahl der Ziel-Virusgruppen und der vorhandenen Nachweis-Methoden werden nach einer gründlichen Literaturanalyse im ersten Jahr erwartet. Nach der Auswertung der Sequenzdaten und dem Primerdesign werden erste Versuche im Labor beginnend im zweiten Jahr durchgeführt. Abhängig von den Ergebnissen, werden die ersten Diagnoseprotokolle im zweiten Jahr erwartet. Zudem werden zeitgleich bereits vorhanden Protokolle im eigenen Labor geprüft und etabliert. Sowohl die Literaturrecherche als auch die Feinjustierung der Protokolle werden bis zu einem zufriedenstellenden Ergebnis fortgeführt.
Projektinformation
Projektleitung: Dr. Veronika Wetzel
Projektbearbeitung: Tunaiye Uapingene
Institut für Pflanzenschutz (IPS)
Phytopathologie und Diagnose, Virologie (IPS 2c)
Lange Point 10, 85345 Freising
Laufzeit: 15.04.2024 bis 31.12.2026
Finanzierung: Bayerisches Staatsministerium für Ernährung, Landwirtschaft, Forsten und Tourismus (StMELF)
Förderkennzeichen: A/23/18