Institut für Pflanzenschutz
Jahresbericht 2021 – Bakteriologie
Bakterien können zahlreiche schwere Krankheiten an Kulturpflanzen auslösen. Eine exakte Diagnose sowie die genaue Identifizierung der Organismen, die die Pflanzenkrankheit ausgelöst haben, ist nicht nur für Bekämpfungs- und Vorbeugungsmaßnahmen von großer Bedeutung. Auch und besonders für die Einhaltung von Regelungen zur Pflanzengesundheit auf EU-Ebene sowie für Ein- und Ausfuhr müssen Krankheitserreger genau identifiziert werden.
An der Bayerischen Landesanstalt für Landwirtschaft ist die Arbeitsgruppe IPS 2b zuständig für die Diagnose bakteriell bedingter Krankheiten an Kulturpflanzen aus Landwirtschaft und Gartenbau.
Nachweis pflanzenpathogener Bakterien im Jahr 2021
| Wirtspflanze | Erreger |
|---|---|
| Bergenie (Bergenia sp.) | Xanthomonas sp. |
| Elsbeere (Sorbus torminalis) | Erwinia amylovora |
| Pelargonie (Pelargonium sp.) | Xanthomonas hortorum pv. pelargonii |
| Platane (Platanus sp.) | Pseudomonas syringae |
| Rosskastanie (Aesculus spp.) | Pseudomonas syringae pv. aesculi |
| Storchschnabel (Geranium sp.) | Xanthomonas sp. |
| Weißdorn (Crataegus spp.) | Erwinia amylovora |
| Wirtspflanze | Erreger |
|---|---|
| Bohne (Phaseolus vulgaris) | Xanthomonas phaseoli |
| Ingwer (Zingiber officinale) | Ralstonia pseudosolanacearum |
| Karotte (Daucus carota) | Candidatus Liberibacter solanacearum |
| Kartoffel (Solanum tuberosum) | Pectobacterium brasiliense, P. parmentieri, Dickeya sp. |
| Liebstöckel (Levisticum officinale) | Pseudomonas syringae, Ps. viridiflava, Xanthomonas sp. |
| Petersilie (Petroselinum crispum) | Pseudomonas syringae, Ps. viridiflava |
| Rote Bete (Beta vulgaris) | Erwinia persicina/ rhapontici |
| Soja (Glycine max) | Pseudomonas savastanoi pv. glycinea, Ps. syringae |
| Sommergerste (Hordeum vulgare) | Pseudomonas syringae pv. atrofaciens |
| Tomate (Solanum lycopersicum) | Pseudomonas syringae |
| Weißkohl (Brassica oleracea capitata var. alba) | Pectobacterium carotovorum ssp. carotovorum, Xanthomonas campestris |
| Zucchini (Cucurbita pepo ssp. pepo | Pseudomonas syringae |
| Zuckerrübe (Beta vulgaris[/i] subsp. vulgaris | SBR (Candidatus Arsenophonus phytopathogenicus) |
| Wirtspflanze | Erreger |
|---|---|
| Apfel (Malus domestica) | Erwinia amylovora |
| Haselnuss (Corylus avellana) | Pseudomonas syringae pv. coryli, Xanthomonas arboricola pv. corylina |
| Kirsche (Prunus avium) | Agrobacterium tumefaciens |
| Pflaume (Prunus domestica) | Pseudomonas syringae pv. mors prunorum |
| Quitte (Cydonia oblonga) | Erwinia amylovora, Pseudomonas syringae |
| Walnuss (Juglans regia) | Pseudomonas syringae, Xanthomonas arboricola pv. juglandis |
| Substrat | Erreger |
|---|---|
| Wasserproben/ Fließgewässer | Ralstonia solanacearum |
Untersuchungen auf geregelte bakterielle Schaderreger
Geregelte Erreger nach EU 2016/2031
Außer regelmäßigen Funden des Schleimfäuleerregers Ralstonia solanacearum in Proben von Fließgewässern (siehe IPS 4b) sowie eines Nachweises von Ralstonia pseudosolancearum an einer Probe von Konsum-Ingwer (siehe unten) konnten in den im Labor eingegangenen Proben keine Unionsquarantäneschädlinge nachgewiesen werden.
 Zoombild vorhanden
Zoombild vorhanden
Xanthomonas arboricola pv. juglandis an Walnuss
Auch Pectobacterium- und Dickeya-Arten an Kartoffeln sind als RNQPs anzusehen, sofern es sich bei dem Material um Pflanzgut handelt. Unabhängig vom tatsächlichen Verwendungszweck der untersuchten Partien wurden auch 2021 zahlreiche Kartoffel-Proben auf Befall mit diesen Schwarzbeinigkeits- bzw. Nassfäuleerregern untersucht. Hierbei handelte es sich in der Regel um Züchtungsmaterial aus frühen Zuchtstufen.
Folgende RNQPs konnten im Jahr 2021 in Proben unterschiedlicher Herkunft nachgewiesen werden:
- Feuerbrand (E. amylovora), mit 26 positiven Befunden bei 107 untersuchten Proben (24%, 2020: 35%)
- Pectobacterium spp. (P. brasiliense, P. atrosepticum, P. parmentieri), mit 31 positiven Befunden bei 74 untersuchten Proben
- Xanthomonas arboricola pv. corylina, mit 51 positiven Befunden bei 66 untersuchten Proben
- Xanthomonas arboricola pv. juglandis, mit 24 positiven Befunden bei 26 untersuchten Proben
- Candidatus Liberibacter solanacearum, mit 10 positiven Befunden bei 25 untersuchten Proben (Möhrensaatgut)
- Pseudomonas syringae pv. morsprunorum an Zwetschge, mit 7 positiven Befunden bei 11 untersuchten Proben
- Xanthomonas phaseoli, mit 2 positiven Befunden bei 3 untersuchten Probe (Saatgut der Schwarzen Bohne)
- Agrobacterium tumefaciens an Kirsche, mit 1 positiven Befund bei 1 untersuchten Probe (siehe unten)
Einige nennenswerte Befunde 2021
Ralstonia pseudosolanacearum an Ingwer
Der Erstfund von R. pseudosolanacearum an Ingwer in Deutschland wurde 2021 aus einem Gewächshaus in Hessen gemeldet. Die dortigen Pflanzen waren aus Ingwerrhizomen gezogen worden, die wahrscheinlich aus Peru für den Konsum importiert worden waren. Es gibt also konkrete Hinweise auf das Risiko einer Einschleppung von R. pseudosolanacearum durch Rhizome von Ingwer (sowie Kurkuma), die eigentlich zum Verzehr eingeführt werden, sofern diese als Pflanzgut genutzt werden.
Auch in Bayern wird seit einigen Jahren von immer mehr Betrieben Ingwer angebaut. Gemäß den Empfehlungen des Julius-Kühn-Instituts sollen an einem Teil der importieren Ware Schnittkontrollen und Latenztestungen bei der Einfuhr durchgeführt werden. Die im Zuge dessen in Bayern untersuchte Ingwer-Charge war komplett für die Verarbeitung (Saftherstellung) bestimmt, so dass im konkreten Fall keine Gefahr einer Ausbreitung des Erregers über Pflanzgut bestand. Anbauer müssen sich jedoch des möglichen Risikos eines Anbaus von Konsum-Ware bewusst sein.
Agrobacterium tumefaciens an Kirschbäumen
 Zoombild vorhanden
Zoombild vorhanden
Agrobacterium tumefaciens an Kirsche
Bodenbakterien der Gattung Agrobacterium sind recht häufig in Böden vorhanden, nicht alle Stämme von A. tumefaciens sind jedoch virulent. Virulente Stämme sind als Träger eines Tumor-induzierenden Plasmids mittels PCR-Methoden verhältnismäßig leicht zu diagnostizieren.
Als Eintrittspforten für A. tumefaciens dienen Wunden durch Pflege- und Kulturmaßnahmen oder durch mechanische Einwirkungen wie Wind, Hagel, Frost oder Tierfraß. Da das Bakterium im Boden überdauert, sind meist die Wurzeln oder Pflanzenorgane in Bodennähe betroffen – daher auch der Name "Wurzelkropf" für diese Krankheit.
Projekt zu SBR (Syndrome Basses Richesses) der Zuckerrübe
 Zoombild vorhanden
Zoombild vorhanden
Blattvergilbung durch SBR bei Zuckerrübe
Der Erreger wird von der Schilf-Glasflügelzikade übertragen, deren Lebenszyklus stark an landwirtschaftliche Fruchtfolgen von Zuckerrüben und Winterweizen angepasst ist. Bisher sind in der Praxis keine wirksamen Bekämpfungsmöglichkeiten bekannt. Die Zikaden sind mit der klassischen Applikation von Blattinsektiziden nicht ausreichend zu bekämpfen. Das Bayerische Staatsministerium für Ernährung, Landwirtschaft und Forsten (StMELF) hat daher ein Forschungsprojekt an der LfL zur Entwicklung von Bekämpfungsstrategien gefördert, bei dem in Zusammenarbeit mit dem Verband Fränkischer Zuckerrübenbauer e.V. alternative Methoden und ackerbauliche Verfahren untersucht werden sollen.
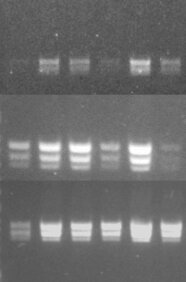 Zoombild vorhanden
Zoombild vorhanden
Optimierung der DNA-Extraktion
Qualitätsmanagement, Akkreditierung und Verbesserung von Nachweismethoden
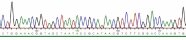 Zoombild vorhanden
Zoombild vorhanden
DNA-Sequenz zur Schaderregeridentifizierung
Im Sinne des Qualitätsmanagements ist es entscheidend, dass die eingesetzten Verfahren hinreichend validiert und verifiziert werden, und sich somit als geeignet erweisen, den gesuchten Schaderreger unter den spezifischen Bedingungen in unserem Labor eindeutig zu diagnostizieren. Insbesondere molekularbiologische Tests (PCR-gestützte Tests, u.U. in Kombination mit Sequenzierverfahren) ermöglichen heute die spezifische und eindeutige Diagnose fast sämtlicher relevanter Schaderreger und werden nach und nach in unserem Labor etabliert und validiert.





